肽鉴定在蛋白质组学中具有关键意义,是理解蛋白质功能和动态的核心。然而,传统数据库搜索方法依赖启发式评分函数,需借助统计估计提高识别率,存在一定局限性。于永瀚和李明院士在 Nature Machine Intelligence(简称NMI,最新影响因子为18.8)上发表文章 Towards highly sensitive deep learning-based end-to-end database search for tandem mass spectrometry 提出 DeepSearch,一种全新的基于深度学习的端到端数据库搜索方法,为质谱肽鉴定提供了革命性的解决方案。
本论文介绍了 DeepSearch,一种基于深度学习的串联质谱端到端数据库搜索方法。DeepSearch 利用对比学习框架下改进的基于变压器的编码器-解码器架构。与依赖离子对离子匹配的传统方法不同,DeepSearch 采用数据驱动的方法来对肽谱匹配进行评分。DeepSearch 还可以以零样本方式分析可变的翻译后修饰。我们表明 DeepSearch 的评分方案表达的偏差较小,并且不需要任何统计估计。我们在各种数据集中验证了 DeepSearch 的准确性和稳健性,包括来自蛋白质组成多样的物种的数据集和富含修饰的数据集。DeepSearch 为串联质谱中的数据库搜索方法提供了新的启示。
在生命科学的前沿,质谱是一种强大的工具,帮助科学家解析蛋白质的“拼图”。在过去8年来,科学界一直面临一个未解的难题:如何更高效、更准确地在海量的数据库中找到肽段的“匹配”。传统的方法效率低,还需要复杂的统计修正,成为蛋白组学探索中的一大瓶颈。
DeepSearch不仅为 π-HuB 项目(人体蛋白质组导航国际大科学计划)提供了质谱数据处理的高效解决方案,还在蛋白质功能动态网络解析、多模态数据融合、高通量蛋白质组分析等领域为 π-HuB 的研究方向和技术实现提供了核心支持。总的来说,尤其在绘制动态蛋白质组图谱和多模态数据整合与智能分析研究两大方向,DeepSearch 的方法推动了π-HuB 的研究进展。这种技术衔接和思路借鉴展示了深度学习如何推动蛋白质组学进入大规模、精细化和精准化的新时代。
DeepSearch 借助对比学习框架,结合改进的变压器编码器-解码器架构,不再依赖离子对离子匹配,而是通过数据驱动的方式实现肽谱匹配评分。其优势包括:
1.零样本能力:可灵活分析多种翻译后修饰(PTMs);
2.偏差更小:无需额外的统计估计,减少了评分方案中的表达偏差;
3.高准确性与稳健性:在包含多物种蛋白质组成的复杂数据集和修饰富集数据集中均展现优异表现。
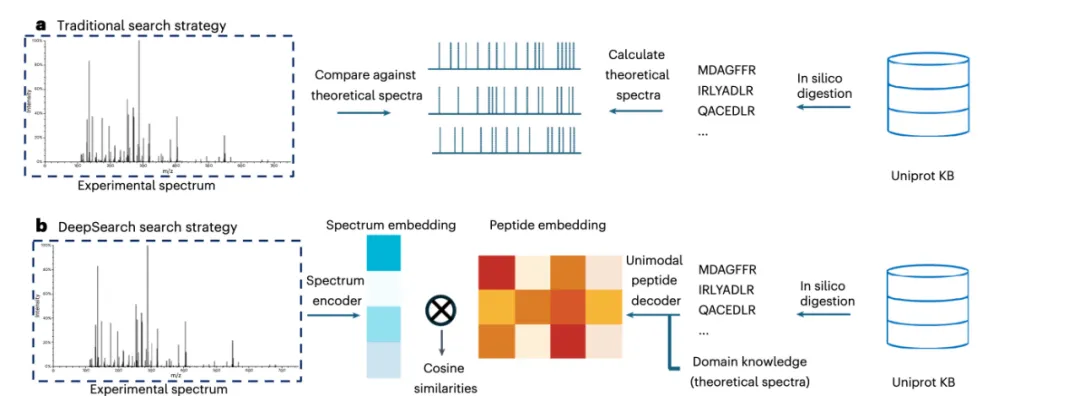
图1.常规数据库搜索通过对比实验光谱和虚拟生成的肽段光谱,找到最匹配的结果。
图2. DeepSearch 利用数值表示和快速数学计算,直接比较实验光谱和肽段的相似性,大幅提升效率。
DeepSearch 改进了传统方法,通过将光谱和肽段转化为数值嵌入表示,并通过矩阵计算快速比较它们的相似性,提升了效率和准确性。
DeepSearch 的创新不仅提升了质谱肽鉴定的效率与精确性,还为探索翻译后修饰和未识别肽提供了强大支持。这标志着质谱数据库搜索技术迈向更智能、更灵活的未来。这一突破标志着质谱数据库搜索技术迈入全新时代,还为探索蛋白质的未知领域打开了全新的大门。
传统方法依赖“对照表”,需要逐个对比数据;而 DeepSearch 的“AI大脑”通过深度学习,像人类直觉一样迅速判断肽谱的匹配情况,完全告别了繁琐的统计估计。
过去 8 年来,这个问题始终没有解决方案。DeepSearch 的诞生,不仅填补了这一空白,还为质谱领域引入了全新的数据驱动方法,从根本上改变了研究的方式。
DeepSearch 不仅能识别普通肽,还能“零样本”检测各种复杂的翻译后修饰。这些修饰往往是生物功能的关键,却在传统方法中难以发现。
参考文献:Yonghan Yu,&Ming Li (2024). Towards highly sensitive deep learning-based end-to-end database search for tandem mass spectrometry,Nature Machine Intelligence . 


